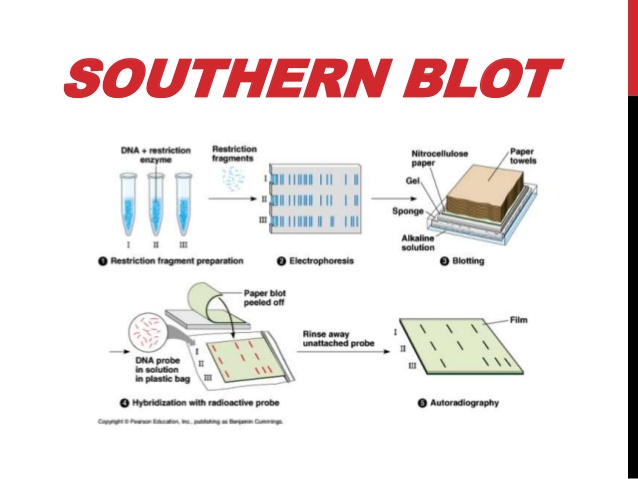
De 106 muestras de heces recolectadas de las provincias de Esmeraldas y Pichincha el 100% fueron positivas para E. histolytica/dispar por microscopía óptica. Por otro lado, al extraer ADN usando QlAamp kit taburete para realizar una Amplificación de ADN mediante PCR en tiempo real (RT-PCR), los ciclos consistieron en:
- 3 minutos a 95°C seguidos de 40 ciclos de:
- 🠉Desnaturalización: 15 segundos a 95°C
- 🠋Hibridación: 30 segundos a 60°C
- 🠉Extensión: 30 segundos a 72°C
La eficacia de la reacción de amplificación se comprobó mediante un corrido electroforético para analizar estos amplicones, se utilizó la secuencia de gen ARNr 18S con fragmentos específicos para E. histoytica (439bp) y E. dispar (174bp). El análisis de datos se realizó con el DA7600 RT PCR y los resultados fueron 74 positivos para E. dispar, 3 positivos para E. histolytica, y 29 fueron negativas para E. histolytica o E. dispar, pudiendo tratarse de E. mosksvskii.
Bibliografía:
1. Guevara A, Vicuña Y, Costales D. Uso de la reacción en cadena de la polimerasa en tiempo real para diferenciar entre Entamoeba histolytica patógena y Entamoeba dispar no patógena en Ecuador [Internet]. 2019 [citado el 23 de diciembre d 2021]. Disponible en: https://pubmed.ncbi.nlm.nih.gov/30398142/
2. Rivero Z, Villarreal L, Bracho A. Identificación molecular de Entamoeba histolytica, Entamoeba dispar y Entamoeba moshkovskii en niños con diarrea en Maracaibo, Venezuela [Internet]. 2021 [citado el 23 de diciembre de 2021]. Disponible en: https://revistabiomedica.org/index.php/biomedica/article/view/5584/4866

No hay comentarios:
Publicar un comentario