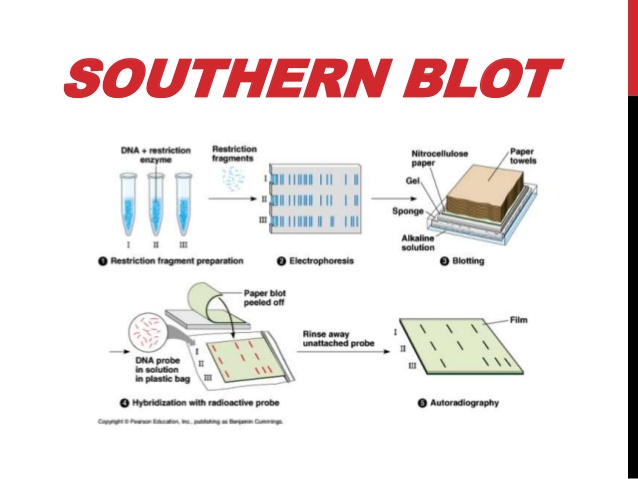
Para el análisis se recolectó alrededor de 5g de heces frescas. Para identificar las cepas, se utilizaron cebadores diseñados a partir de las secuencias flanqueantes de diferentes copias de EhSINE1 y EhSINE2, obtenidas de la base de datos E. histolytica HM-1:IMSS. Después, el cebador SINE se ejecutó en gel de agarosa y se transfirió a una membrana GeneScreen. Luego, las sondas marcadas radiactivamente se prepararon utilizando α-32PedATP mediante cebado aleatorio.
HIBRIDACIÓN
1.- Se hizo durante la noche a 65 °C en tampón de hibridación con sondas de ADN específicas de locus.
2.- La membrana fue lavada dos veces a temperatura ambiente en 2x SSC.
3.- Seguido de 2x SSC y SDS al 1% a 65 °C durante 7 minutos.
4.- Dos veces expuesta con 0,1x SSC durante 20 minutos a temperatura ambiente.
5.- Por último, fue escaneada con fósforo reproductor de imágenes.
De 187 muestras analizadas, 70 muestras fueron positivas para E. histolytica o E. dispar, basados en los loci polimórfico EhSINE1 y EhSINE2.
Bibliografía:
1. Sharma S, Banyal N, Singh M, Mandal A, Bhattacharya S. El polimorfismo SINE revela distintas cepas de Entamoeba histolytica del norte de la India [Internet]. Parasitología Experimental. 2017 [citado el 09 de enero de 2022]. Disponible en: https://www.sciencedirect.com/science/article/abs/pii/S0014489416302302?via%3Dihub